
李国民

特聘研究员
gmli(at)cimrbj.ac.cn
武汉大学 生物学系 学士
武汉大学 生物学系 硕士
美国韦恩州立大学 化学系 博士
工作经历
2023至今
首都医学科学创新中心 肿瘤研究所 特聘研究员
2023至今
首都医科大学 讲席教授
2017.6-2022
美国得克萨斯大学西南医学中心 讲席教授
2017.6-2022
美国得克萨斯大学西南医学中心 Reece A. Overcash Jr.结肠癌研究中心 主任
2017.6-2022
美国得克萨斯大学西南医学中心放射肿瘤科 转化研究主任
2015.8-2017.5
美国南加州大学医学院生物化学与分子生物学系 讲席教授
2006-2015.7
美国肯塔基大学毒理和肿瘤生物学系 讲席教授
2000-2005
美国肯塔基大学病理学系 长聘副教授
1995-1999
美国肯塔基大学病理学系助理教授
研究方向
李国民实验室研究 DNA 错配修复 (DNA mismatch repair, MMR) 机理及其在肿瘤和神经退行性疾病发生、发展、治疗中的作用。
主要研究课题
1. DNA错配修复的机制研究
DNA错配修复(MMR)通过纠正新合成链中的复制错误来维持基因组稳定。我们离体重塑MMR的研究表明,欧博MutS和MutL家族蛋白识别错配,起动错配修复反应,二者招募核酸酶 Exo1来切除错配碱基。所产生的缺口由DNA聚合酶填补,从而完成醋配修复反应(见下图)。尽管进行了大量研究,MMR中许多基本问题仍未解决。比如:
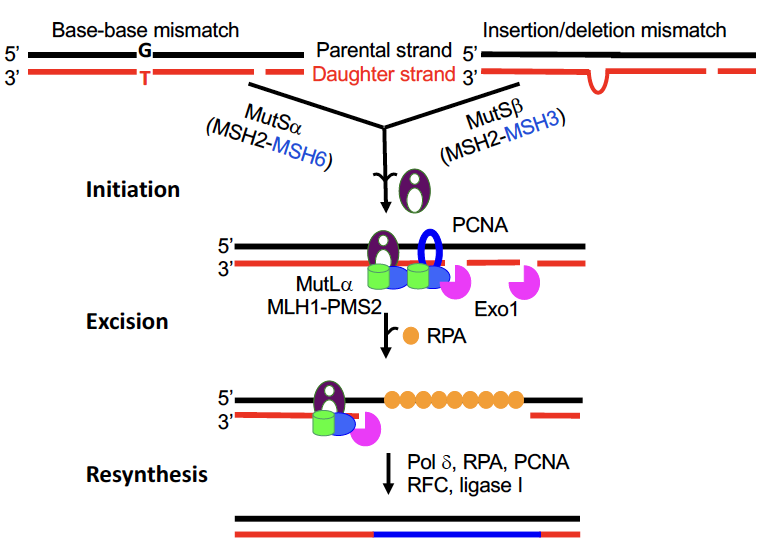
1)MMR是如何特异性地定位到新合成的链上? 与其他DNA修复途径要修复的DNA损伤不同,欧博娱乐MMR要修复的错配碱基(例如,GT或CA)都是正常的DNA组分。因此,为了去除错配的碱基,MMR系统必须知道哪条链是子链。早起的研究表明,新合成的子链含有DNA缺口,欧博allbet可作为链鉴别信号,以确保 MMR定位到子链上。然而,缺口通常与错配相距几百个碱基,MMR系统如何在这两个位点进行通信一直是该领域的一个悬而未决的谜题。我们将利用MMR 的重塑系统来解决这一难题。
2)MMR在体内是如何发生的? 我们对MMR机制的了解主要来自使用裸DNA的体外研究。然而,在体内,欧博百家乐DNA被包装成由核小体组成的染色质。MMR在体内是如何发生的仍不明确。虽然我们早期实验室表明,人类错配识别蛋白MutSa通过与H3K36me3(组蛋白H3赖氨酸36三甲基化)的相互作用被招募到染色质上,但其他MMR蛋白是如何被招募以及它们在体内是如何相互作用的尚待研究。这是我们要解的另一难题,它将为癌症诊断和治疗提供关键信息。

2. 基于错配修复缺陷的癌症靶向治疗
尽管经过数十年的研究,癌症仍然是主要的死因之一。其主要问题在于没有有效根治肿瘤的药物。最近的研究表明,MMR缺陷肿瘤对免疫治疗的反应很好,但其机制尚不明了。我们实验室正在研究MMR缺陷如何引发免疫治疗的机制,并开发方法来降低或阻断癌细胞的MMR活性,以便所有癌都可以适用于免疫治疗。

3. 错配修复在神经退行性疾病中的作用
三核苷酸重复序列的扩增引起30多种严重的神经退行性疾病,包括亨廷顿病、肌萎缩性脊髓侧索硬化症和脆性X综合症。虽然MMR系统以维持DNA复制的准确性著称,但MutSβ醋配识别蛋白是促进三核苷酸重复序列扩增的关键因素。我们正在研究其机理和开发、筛选抑制MutSβ活性的小分子,从而抑制三核苷酸重复序列的扩增和治疗神经退行性疾病。

4. 解析错配修复起始复合物的结构
晶体结构和低温电子显微镜(cryo-EM)研究已经解析了许多蛋白质的结构,包括一些单个MMR蛋白的结构。 错配修复起始复合物(包含错配DNA、MutSa、MutLa、PCNA、RPA和Exo1)的形成是MMR系统的关键步骤,是理解 MMR 机理的重要反应。但迄今为止错配修复起始复合物的结构尚未解析。这将是我们要攻克的另一难题。错配修复起始复合物结构的解析不仅提供MMR的反应机制,还将为开发癌症治疗药物提供策略。

主要成果与贡献
1. 发现 MMR 缺陷导致多种遗传性和散发性癌 (Cell,1993;Science,1995)

2. 体外重塑人类 MMR 反应 (Cell,2005;Cell Research,2021)

3. 发现表观遗传因子和组蛋白标记物 H3K36me3 为重要MMR组分 (Cell,2013)

4. 阐明 MMR 缺陷促进肿瘤免疫检点疗法机制 (Cancer Cell,2021a;Cancer Cell,2021b)

5. 阐明促进DNA (CAG) (CTG)重复序列扩增导致多种神经退行性疾病机理 (Cell Research,2016)

代表性文章
代表性文章
Sun L, Liu L, Wu C, Li X,, Guo J, Zhang J, Guan J, Wang N, Gu L, Yang XW, Li GM. Mutant huntingtin protein induces MLH1 degradation, DNA hyperexcision, and cGAS–STING-dependent apoptosis. Proc Natl Acad Sci U S A. , 2024, 121:e2313652121. DOI: 10.1073/pnas.2313652121
Huang Y, Gu L, Li GM. Heat shock protein DNAJA2 regulates transcription-coupled repair by triggering CSB degradation via chaperone-mediated autophagy. Cell Discovery, 2023, 9: 107. DOI: 10.1038/s41421-023-00601-8
Huang Y* , Lu C*, Wang H, Gu L, Fu YX#, Li GM#. DNAJA2 deficiency activates cGAS-STING pathway via the induction of aberrant mitosis and chromosome instability. Nature Communications, 2023, 14: 5246. DOI: 10.1038/s41467-023-40952-0
Zhang J, Zhao X, LiuL, Li HD, Castrillon DH, Gu L, Li GM. The mismatch recognition protein MutSa promotes nascent strand degradation at stalled replication forks. Proceedings of the National Academy of Sciences of the United States of America. PNAS, 2022, 119: e2201738119. DOI: 10.1073/pnas.2201738119
Guan J*, Lu C*, Jin Q, Lu H, Chen X, Tian L, Zhang Y, Ortega J, Zhang J, Siteni S, Chen M, Gu L, Shay J, Davis A, Chen ZJ, Fu YX#, Li GM#. MLH1 deficiency-triggered DNA hyperexcision by exonuclease1 activates the cGAS-STING pathway. Cancer Cell, 2021, 39: 109-121. DOI: 10.1016/j.ccell.2020.11.004
Lu C*, Guan J*, Lu S, Jin Q, Rousseau B, Lu T, Stephens D, Zhang H, Zhu J, Yang M, Ren Z, Liang Y, Liu Z, Han C, Liu L, Cao X, Zhang A, Qiao J, Batten K, Chen M, Castrillon DH, Li B, Li GM#, Fu YX#. DNA sensing in mismatch repair-deficient tumor cells is essential for anti-tumor immunity. Cancer Cell, 2021, 39: 96-108. DOI: 10.1016/j.ccell.2020.11.006
Ortega J*, Lee GS*, Gu L, Yang W, Li GM. Mispair-bound human MutS-MutL complex triggers DNA incisions and activates mismatch repair. Cell Research, 2021, 31: 542-553. DOI: 10.1038/s41422-021-00468-y
Huang Y*, Zhao J*, Mao G, Lee GS, Zhang J, Bi L, Gu L, Chang Z, Valentino J#, Li GM#. Identification of novel genetic variants predisposing to familial oral squamous cell carcinomas. Cell Discovery, 2019, 5: 57. DOI: 10.1038/s41421-019-0126-6
Fang J*, Huang Y*, Mao G*, Yang S, Rennert G, Gu L, Li H, Li GM. Cancer-driving H3G34V/R/D mutations block H3K36 methylation and H3K36me3-MutSa. Proc Natl Acad Sci USA, 2018, 115: 9598-9603. DOI: 10.1073/pnas.1806355115
Guo J, Gu L, Leffak M, Li GM. MutSβ promotes trinucleotide repeat expansion by recruiting DNA polymerase β to nascent (CAG)n or (CTG)n hairpins for error-prone DNA synthesis. Cell Research, 2016, 26: 775-786. DOI: 10.1038/cr.2016.66
Li F, Mao G, Tong D, Huang J, Gu L#, Yang W, Li GM#. The histone mark H3K36me3 regulates human DNA mismatch repair through its interaction with MutSα. Cell, 2013, 153: 590-600. DOI: 10.1016/j.cell.2013.03.025